数据结构实验 - DNA序列匹配
实验过程
任务要求
分别利用BF算法和KMP算法实现DNA序列的匹配.
分析
- 在之前的课程中已经实现了两种匹配算法,只需要稍作修改,改成返回bool值即可.
- 核心思路:
- 遍历生成环状DNA序列;
- 利用算法匹配;
文件分配
dnamatch.cpp: DNA序列匹配算法;match.cpp: 两种匹配算法;utils.cpp: 读入与保持数据;gen_test.py: 生成测试数据;
主函数
1 | int main() |
这段主函数先从test_data/input.csv读入数据,然后利用gen_unique_patterns函数生成所有需要匹配的DNA串,然后利用bf_search函数进行匹配,最后将结果保存到test_data/output.csv中。同时还利用了chrono库进行计时。
gen_unique_patterns函数
对于任意一个环状DNA序列,可以生成n个不同的模式串,但是考虑到可能会产生重复的情形,所以需要生成不重复的模式串。
对于这一要求,我们有两种可选的方案:
- 生成所有的模式串,然后利用
set去重; - 我们可以通过观察发现,只有原始串发生本身重复的情况,才会产生重复的模式串。例如abc就只有abc, bca, cab三种,而对于abab就只有abab与baba两种,其本质是ab与ba的重复。
但是考虑到通常模式串并不会特别大,同时利用set的代码比较方便,所以这里我们直接使用第一种方法。
1 | set<string> gen_unique_patterns(string virus_dna) |
测试函数
生成测试数据
这里撰写了一个简单的python脚本gen_test.py,用于生成一些测试数据。
测试结果
我分别生成了一些测试数据,包括人类DNA序列逐渐增长的,已及病毒DNA逐渐增长的,还有一些模式串自身重复的,与匹配串大量重复的。
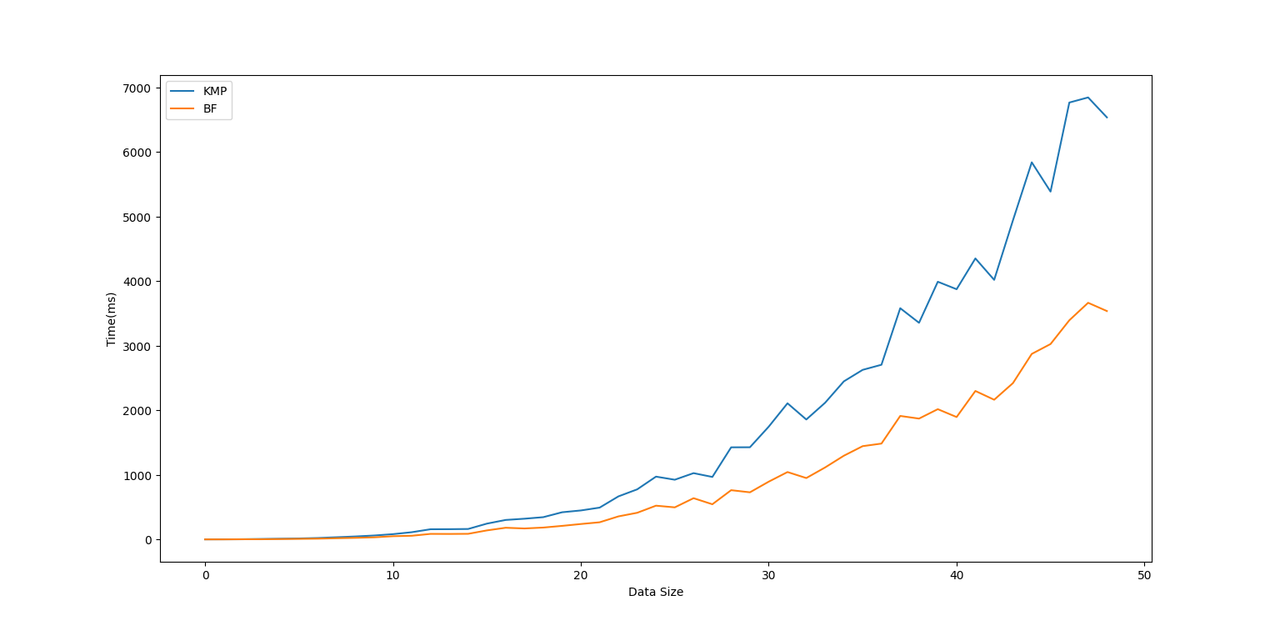
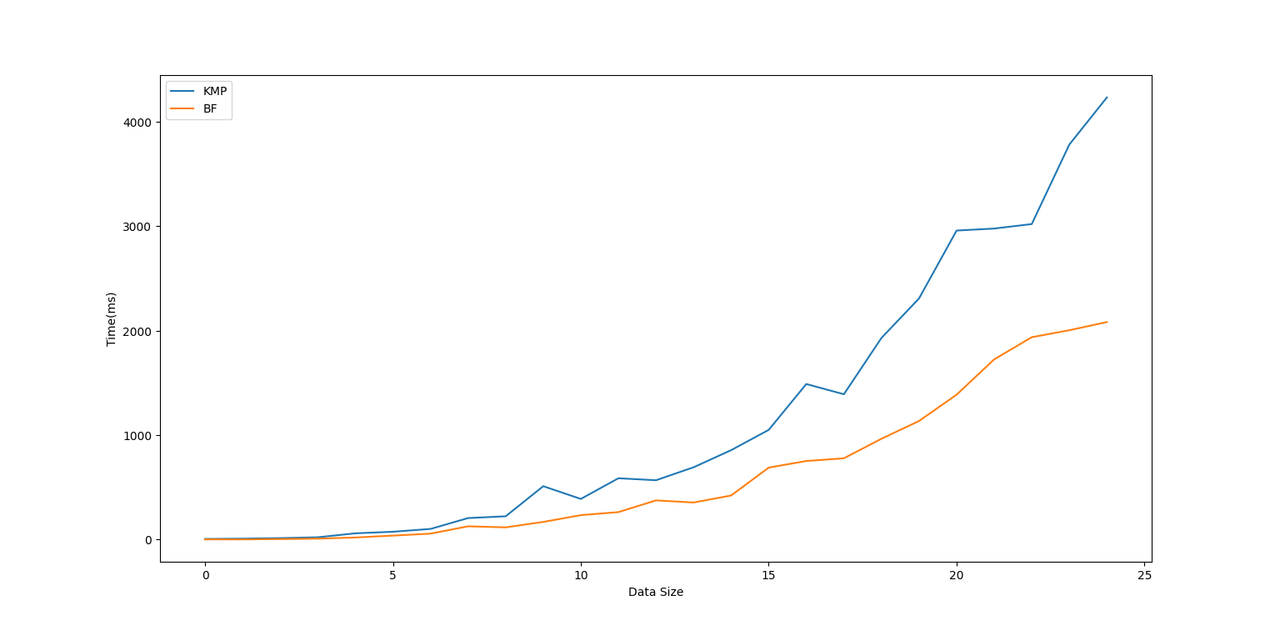
根据这两幅图的运行结果,我们可以很明显的看出,KMP算法的运行时间几乎在BF算法的两倍,这是为什么呢?经过我对代码不同部分的运行时间分析,首先主要是因为我使用了vector来构造lps数组,大量标准库容器的使用虽然方便并且功能强大,但是也带来了一些性能损失。
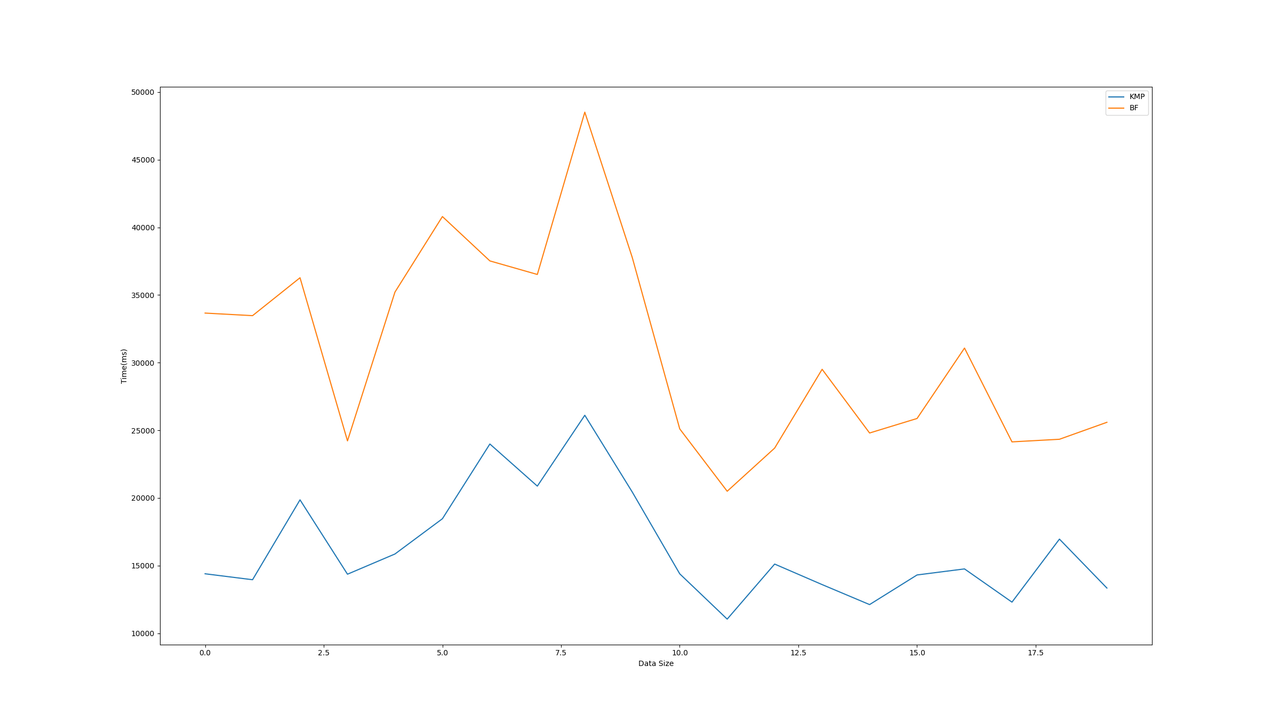
但在这幅图上,形势发生了逆转,BF算法的运行时间几乎变成KMP算法的两倍,这是因为这组数据是专门为KMP算法设计的,模式串本身发生大约15次重复,匹配串则取模式串的前n-1的字符发生10000次重复,从而导致匹配只能发生在匹配串的末尾,并且需要大量的回退操作,因此在这种情况下,lps的计算开销就显得微不足道了,从而让KMP算法的性能优于BF算法。
总结
本实验项目撰写的KMP算法并非性能最优,仅仅只是达到了算法演示的效果,在多数情况下BF算法的性能更优,只有在数据具备特殊特征的情况下 才会出现KMP算法的性能优势。